Détection de la fragmentation de l’ADN
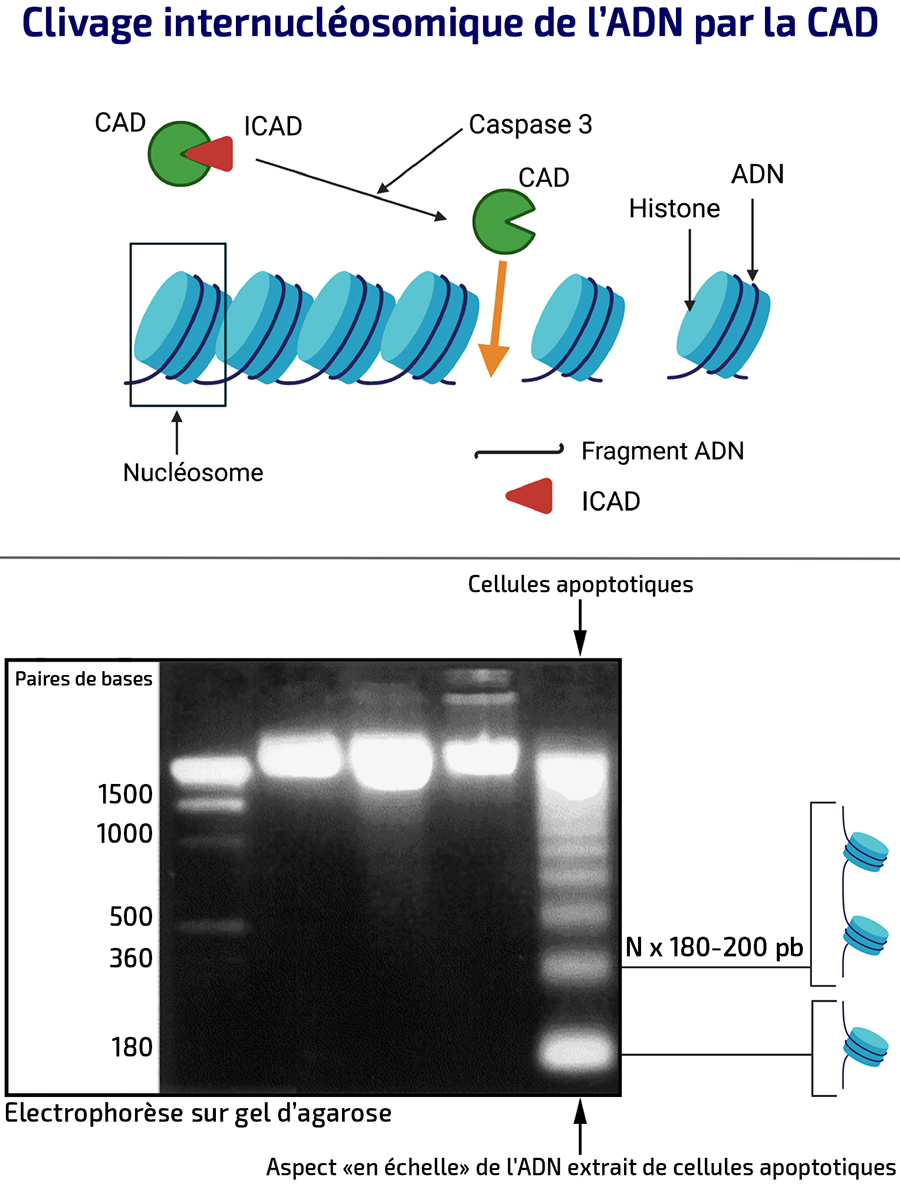
Clivage internucléosomique de l'ADN par la Caspase-Activated-DNase (CAD)
La fragmentation de l’ADN est un événement tardif. Au cours de l’apoptose, l’ADN est hydrolysé spécifiquement en fragments dont les tailles sont des multiples de 180-200 paires de bases. Cette taille est révélatrice de l'espacement entre deux nucléosomes consécutifs. Ce clivage est réalisé par la Caspase-Activated-DNase (CAD) qui existe, dans les cellules saines, sous forme d’un complexe inactif car liée à une sous-unité régulatrice ICAD (Inhibitor of Caspase Activated DNase). Au cours de l’apoptose, le clivage de l’ICAD par la caspase-3 activée conduit à la libération de la CAD qui transloque dans le noyau et clive l’ADN : ce qui aboutit à un clivage régulier de l’ADN internucléosomal (multiples de 180-200 paire de bases).
Détection sur gel d’agarose de l’ADN clivé
Ces fragments dont les tailles sont des multiples de 180-200 paires de bases peuvent être séparés selon leur taille par électrophorèse sur un gel d’agarose après coloration au bromure d’éthidium de l’ADN clivé. Ces fragments apparaissent sous forme d’un profil caractéristique dit « en barreau d’échelle d’ADN » traduisant la fragmentation de l’ADN.
Détection par la méthode TUNEL
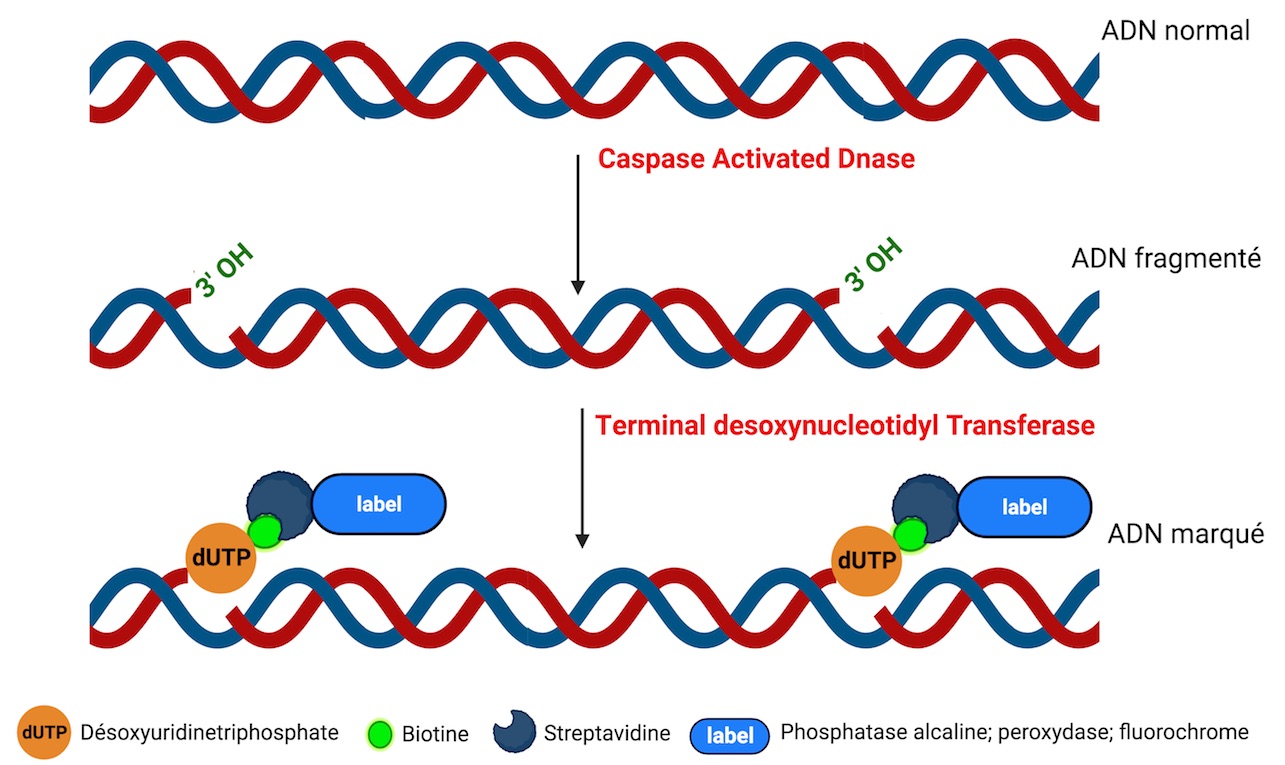
La méthode TUNEL est un marquage qui permet de mettre en évidence in situ la fragmentation de l'ADN nucléaire résultant de l'activation de la Caspase-Activated DNase (CAD) au cours de l'apoptose. Cette méthode est basée sur le marquage des extrémités 3'-OH libres de l'ADN par la Terminal desoxynucleotidyl Transférase (TdT) : cette enzyme catalyse l'accrochage au niveau des extrémités 3'-OH libres de l’ADN de nucléotides triphosphate biotinylés (par example les désoxyuridines triphosphates nucléotides (dUTP) marqués par la biotine). La Biotine est détectée par la Streptavidine, elle même couplée à la phosphatase alcaline, à la peroxydase ou à un fluorochrome. Ainsi, le marquage peut être examiné au microscope à contraste de phase ou à fluorescence ou par cytométrie en Flux en fonction de l’étiquette. Il existe des variantes de la méthode TUNEL en fonction des désoxynucléotides triphosphates utilisés et de leur marquage directe ou indirecte par l’étiquette.
Terminal desoxynucléotidyl transferase dUTP Nick End Labeling
.
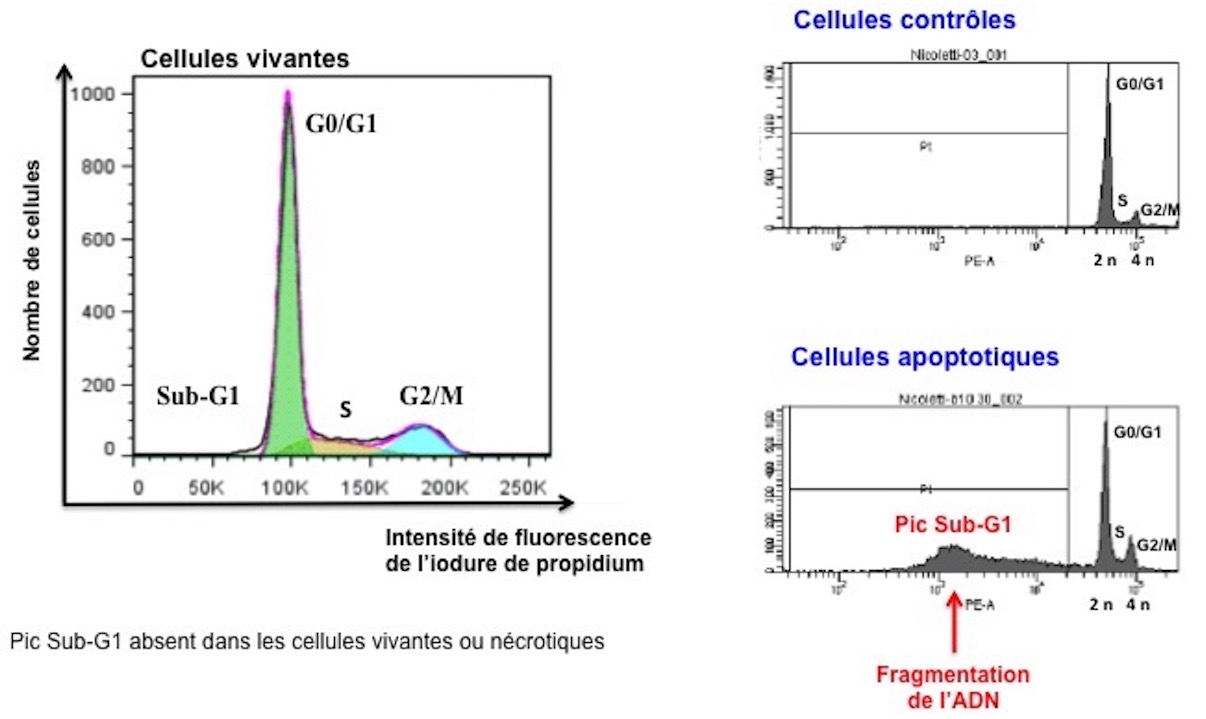
Détection du pic SubG1
La fragmentation de l’ADN peut être également évaluée par cytométrie en flux sur des cellules perméabilisées. Techniquement, la fragmentation de l'ADN est facile à détecter en raison de la perte d'ADN des cellules perméabilisées. Lorsque les cellules sont perméabilisées, par exemple par de l'éthanol à 70 %, les multimères d'ADN fragmentés de 180-200 pb s'échappent de la cellule apoptotique pendant les étapes de lavages. Le résultat est une population de cellules avec une teneur réduite en ADN. Après coloration par l’iodure de propidium, la teneur en ADN des cellules perméabilisées pourra être quantifiée car elle est proportionnelle à l’intensité de fluorescence émise par l’iodure de propidium qui se lie stoechiométriquement aux acides nucléiques. Ainsi des cellules apoptotiques présentent un pic Sub-G1 caractéristique localisé avant le pic G0/G1. En revanche les cellules vivantes ou nécrotiques ne présentent pas de pic Sub-G1.
% Sub-G1 = % cellules apoptotiques = % de cellules avec une fragmentation internucléosomale de l’ADN.